XRCC5/Ku80 Monoklonaler Antikörper
XRCC5/Ku80 Monoklonal Antikörper für WB, IHC, IF/ICC, IP, ELISA
Wirt / Isotyp
Maus / IgG1
Getestete Reaktivität
human, Maus, Ratte
Anwendung
WB, IHC, IF/ICC, IP, ELISA
Konjugation
Unkonjugiert
CloneNo.
2G5E7
Kat-Nr. : 66546-1-Ig
Synonyme
Geprüfte Anwendungen
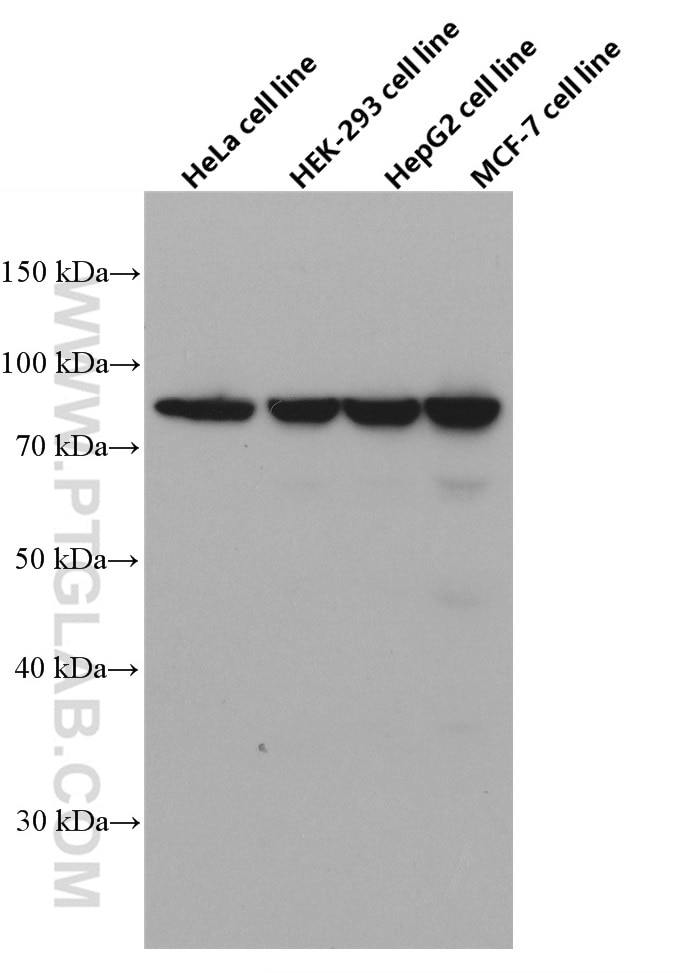
| Erfolgreiche Detektion in WB | HeLa-Zellen, HEK-293-Zellen, HepG2-Zellen, MCF-7-Zellen |
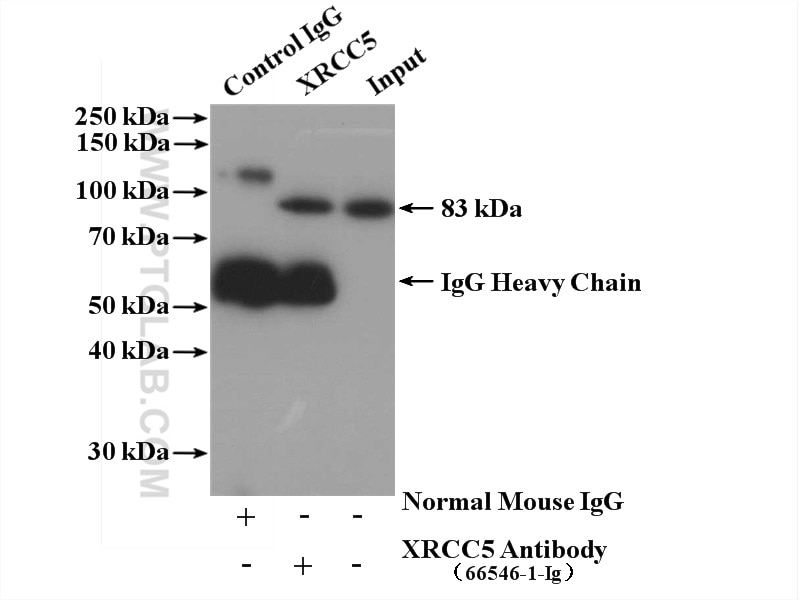
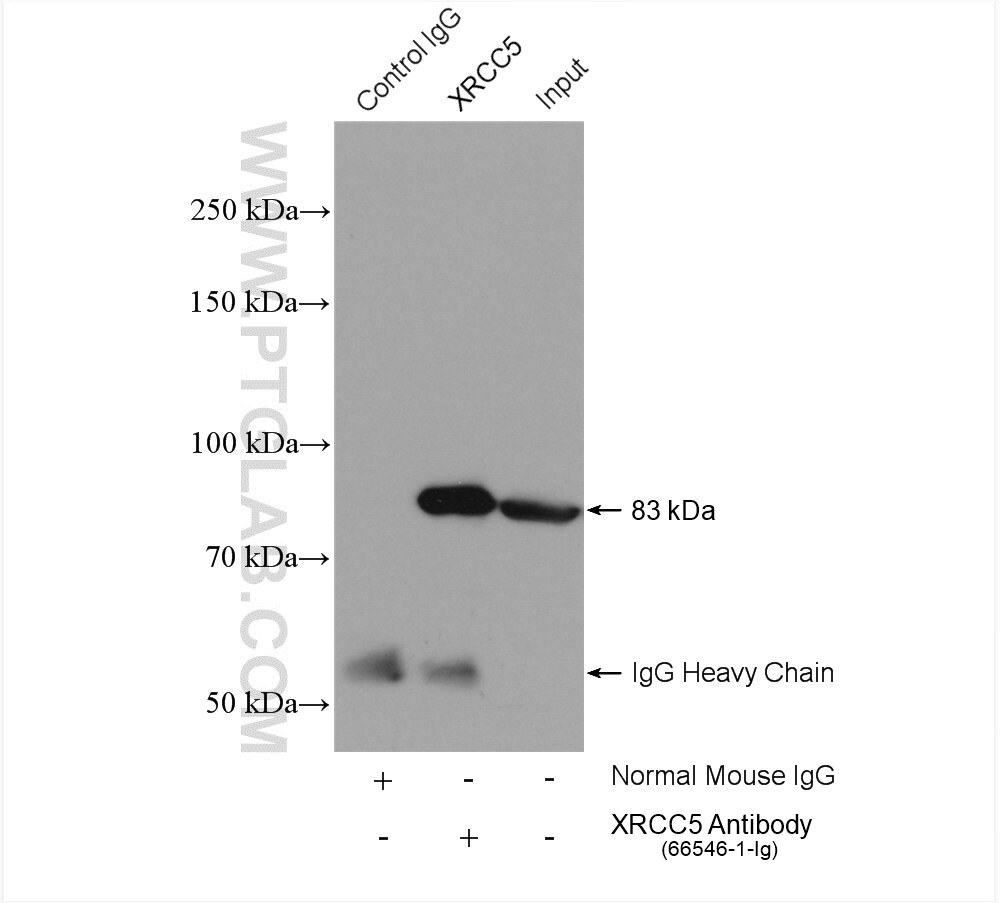
| Erfolgreiche IP | HeLa-Zellen |
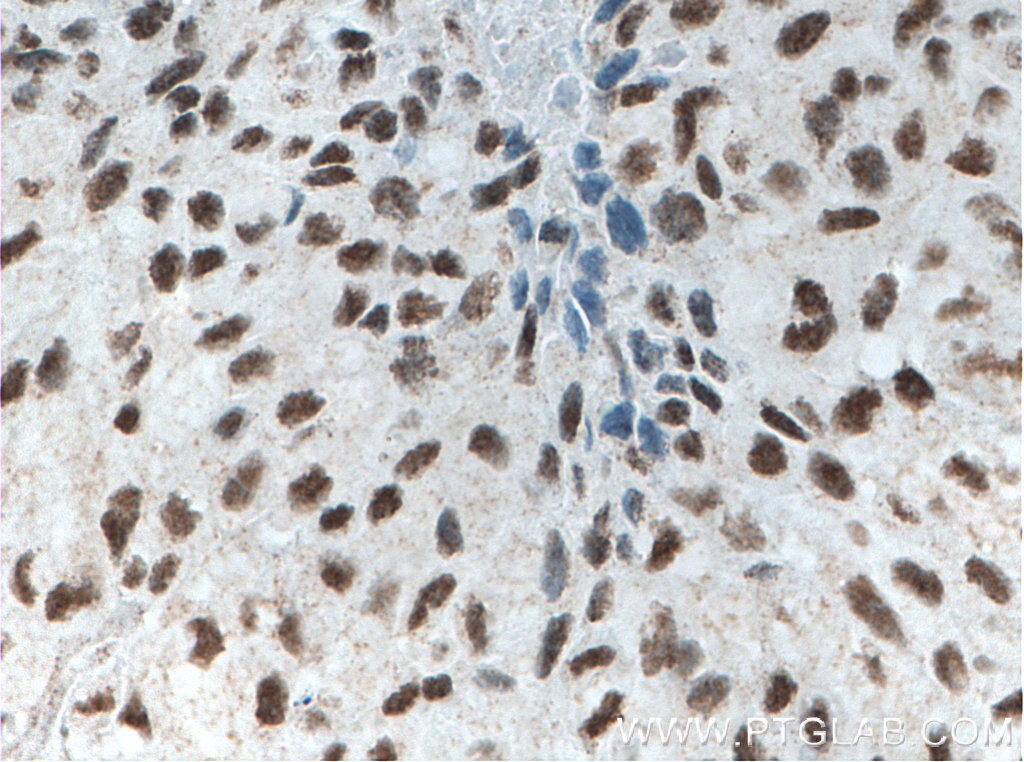
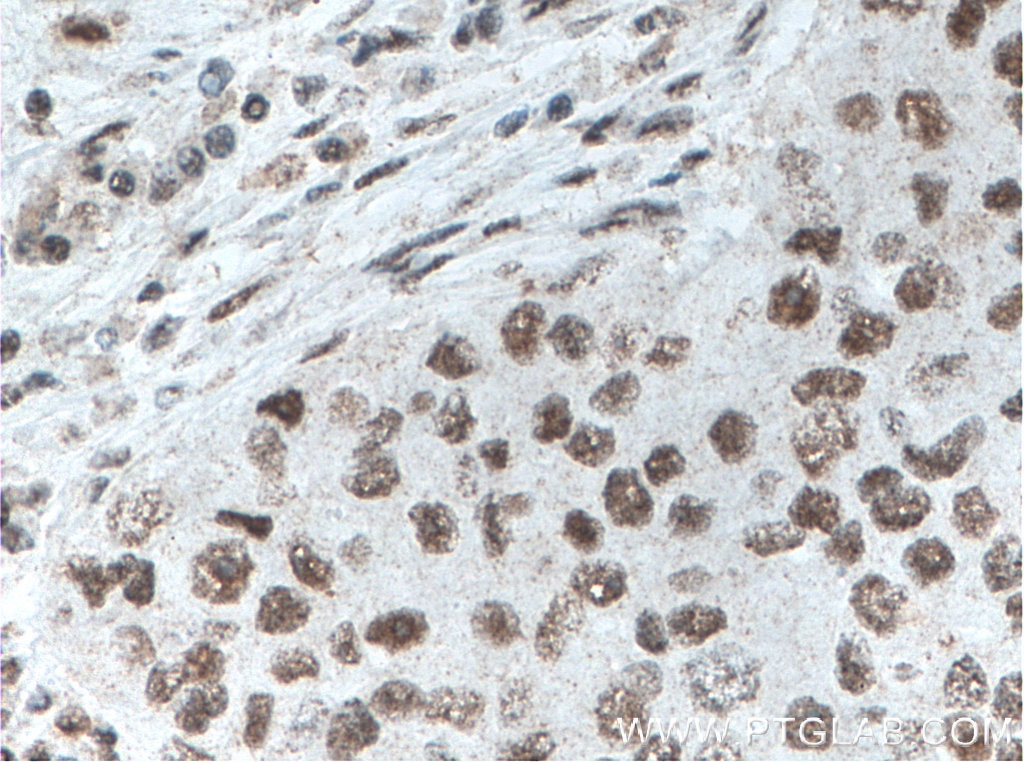
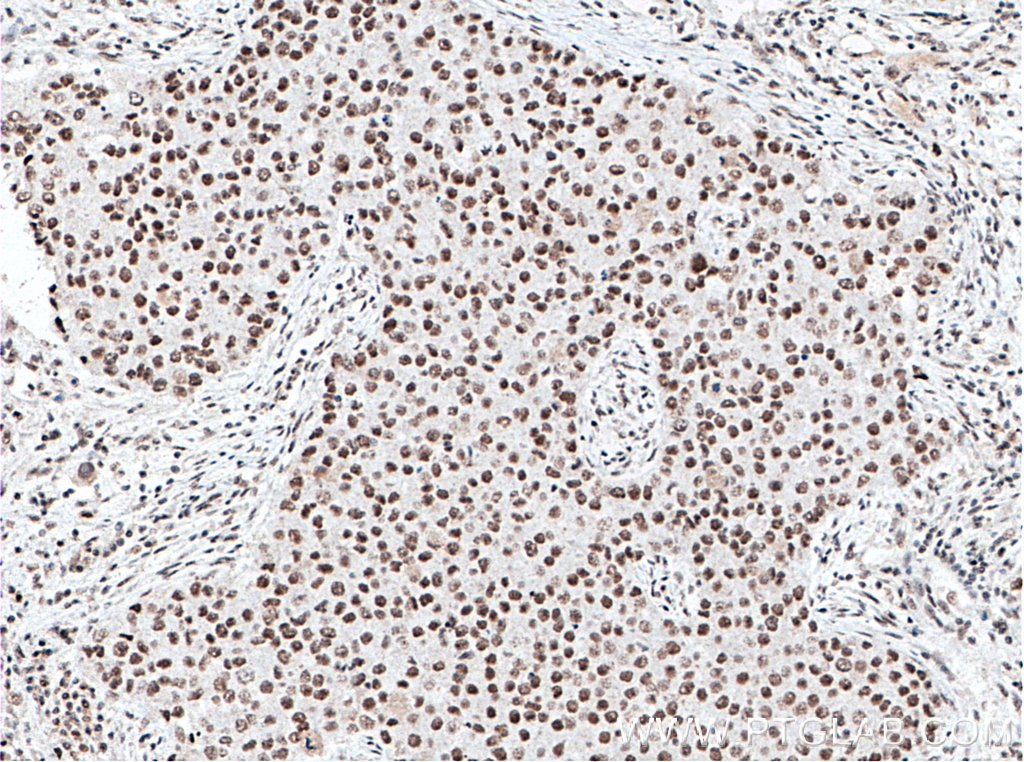
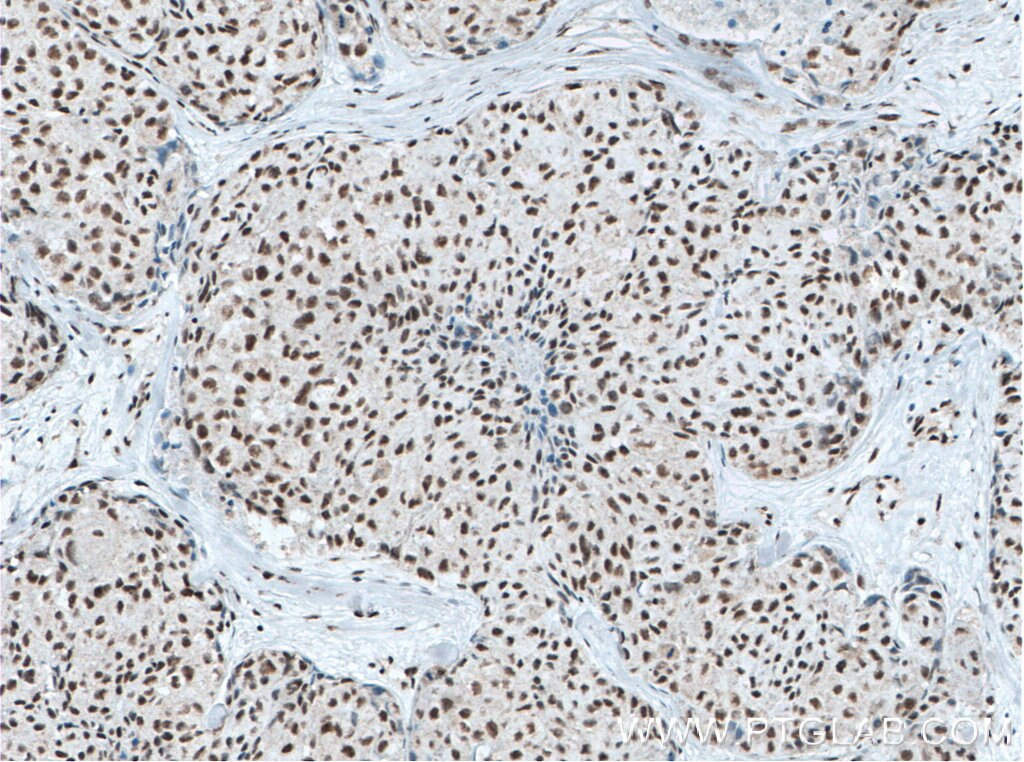
| Erfolgreiche Detektion in IHC | humanes Lungenkarzinomgewebe, humanes Mammakarzinomgewebe Hinweis: Antigendemaskierung mit TE-Puffer pH 9,0 empfohlen. (*) Wahlweise kann die Antigendemaskierung auch mit Citratpuffer pH 6,0 erfolgen. |
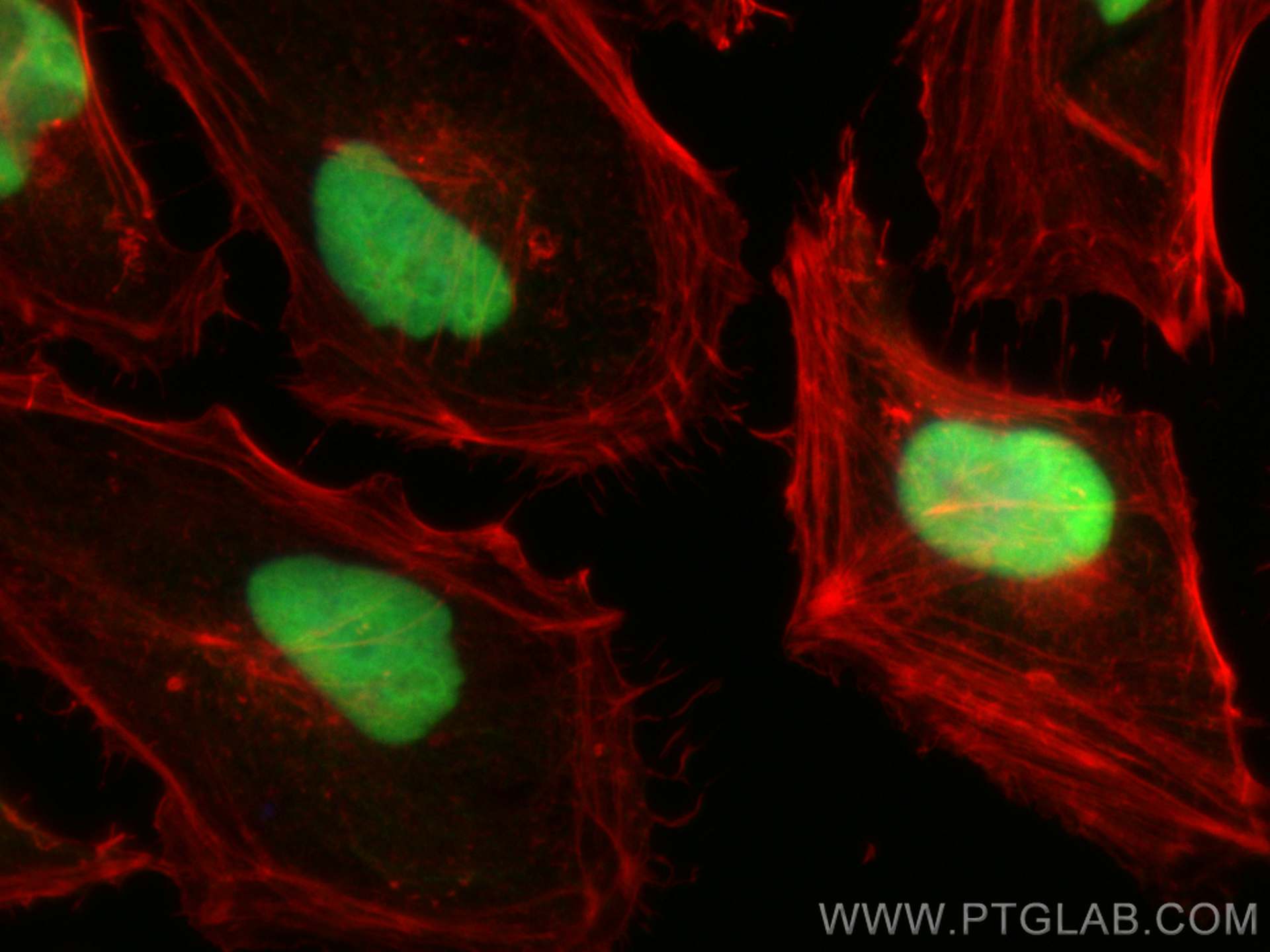
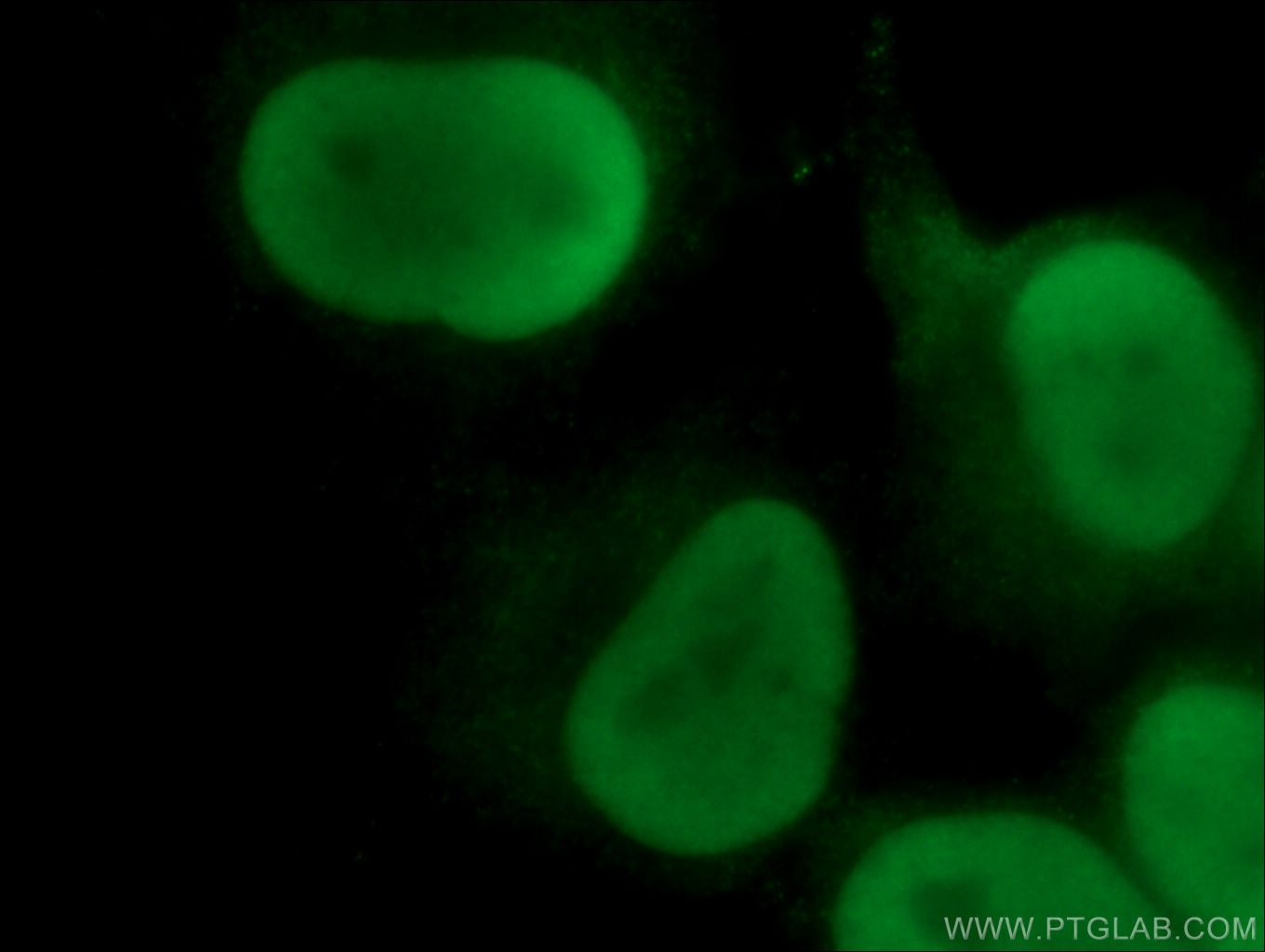
| Erfolgreiche Detektion in IF/ICC | HeLa-Zellen |
Empfohlene Verdünnung
| Anwendung | Verdünnung |
|---|---|
| Western Blot (WB) | WB : 1:5000-1:50000 |
| Immunpräzipitation (IP) | IP : 0.5-4.0 ug for 1.0-3.0 mg of total protein lysate |
| Immunhistochemie (IHC) | IHC : 1:500-1:2000 |
| Immunfluoreszenz (IF)/ICC | IF/ICC : 1:200-1:800 |
| It is recommended that this reagent should be titrated in each testing system to obtain optimal results. | |
| Sample-dependent, check data in validation data gallery | |
Veröffentlichte Anwendungen
| WB | See 5 publications below |
| IF | See 4 publications below |
Produktinformation
66546-1-Ig bindet in WB, IHC, IF/ICC, IP, ELISA XRCC5/Ku80 und zeigt Reaktivität mit human, Maus, Ratten
| Getestete Reaktivität | human, Maus, Ratte |
| In Publikationen genannte Reaktivität | human, Ratte |
| Wirt / Isotyp | Maus / IgG1 |
| Klonalität | Monoklonal |
| Typ | Antikörper |
| Immunogen | XRCC5/Ku80 fusion protein Ag9512 |
| Vollständiger Name | X-ray repair complementing defective repair in Chinese hamster cells 5 (double-strand-break rejoining) |
| Berechnetes Molekulargewicht | 732 aa, 83 kDa |
| Beobachtetes Molekulargewicht | 80-83 kDa |
| GenBank-Zugangsnummer | BC019027 |
| Gene symbol | XRCC5 |
| Gene ID (NCBI) | 7520 |
| Konjugation | Unkonjugiert |
| Form | Liquid |
| Reinigungsmethode | Protein-G-Reinigung |
| Lagerungspuffer | PBS with 0.02% sodium azide and 50% glycerol |
| Lagerungsbedingungen | Bei -20°C lagern. Nach dem Versand ein Jahr lang stabil Aliquotieren ist bei -20oC Lagerung nicht notwendig. 20ul Größen enthalten 0,1% BSA. |
Hintergrundinformationen
There are at least two pathways for eukaryotes to repair DNA double-strand breaks: homologous recombination and nonhomologous end joining(NHEJ). The core NHEJ machinery includes XRCC4, DNA ligase IV and the DNA-dependent protein kinase complex, which consists of the DNA end-binding XRCC5/XRCC6 heterodimer and the catalytic subunit PRKDC. The heterdimer of XRCC5/XRCC6 enhanced teh affinity of the catalytic subunit PRKDC to DNA by 100-fold. Once the XRCC5/6 dimer association with NAA15, it can bind to the osteocalcin promoter and activate osteocalcin expression. The XRCC5/6 dimer acts as a negative regulator of transcription when together with APEX1. Some publised papers indicated that the MW of XRCC5 is 86kDa, while more papers suggested that XRCC5 is a 80kDa protein, as it was firstly introducted in publication. Thus, Ku80 and Ku86 are the same protein.
Protokolle
| PRODUKTSPEZIFISCHE PROTOKOLLE | |
|---|---|
| WB protocol for XRCC5/Ku80 antibody 66546-1-Ig | Protokoll herunterladen |
| IHC protocol for XRCC5/Ku80 antibody 66546-1-Ig | Protokoll herunterladenl |
| IF protocol for XRCC5/Ku80 antibody 66546-1-Ig | Protokoll herunterladen |
| IP protocol for XRCC5/Ku80 antibody 66546-1-Ig | Protokoll herunterladen |
| STANDARD-PROTOKOLLE | |
|---|---|
| Klicken Sie hier, um unsere Standardprotokolle anzuzeigen |
Publikationen
| Species | Application | Title |
|---|---|---|
Cancer Lett ITGA2 overexpression inhibits DNA repair and confers sensitivity to radiotherapies in pancreatic cancer | ||
DNA Repair (Amst) FAM21 interacts with Ku to promote the localization of WASH to DNA double strand break sites | ||
Invest New Drugs Radiosensitization effect of quinoline-indole-schiff base derivative 10E on non-small cell lung cancer cells in vitro and in tumor xenografts | ||
FEBS Open Bio Establishment and biological characterization of radioresistant colorectal cancer cell lines |